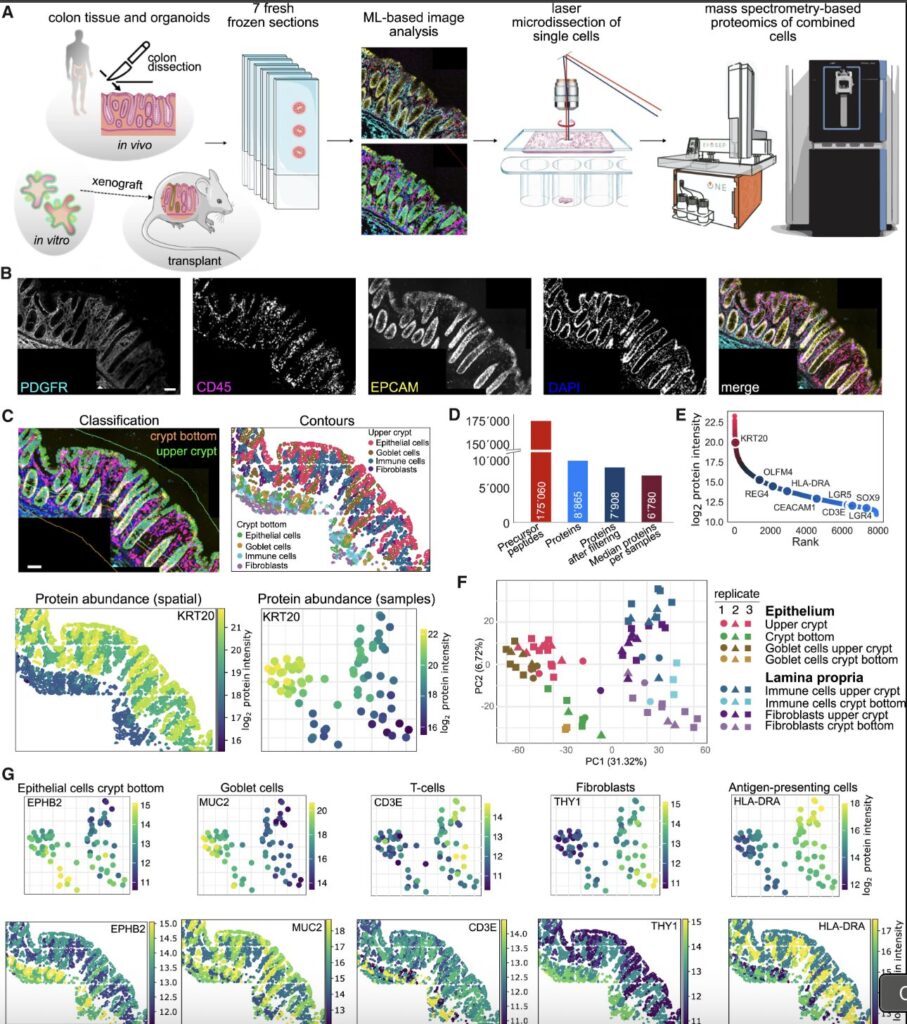
切片画像からセルセグメンテーションを行い、その座標をLMD(レーザーマイクロダイセクション)に読み込ませ切り取り→LC-MSにて測定することで、single cell レベルの空間プロテオミクスを実現した論文。
Matthias Mann labからは最近この手の論文が複数出てきていますね。空間情報を保持したままsingle cellレベルのデータを取得するのはまさに究極の域に達していると思います。
Evosep, Orbitrap Astral, LMD7という高額のハードだけでなくソフトウェア面でもなかなか導入ハードルは高そうです。。。
LMDでsingle cellレベルで切り出し、それぞれから5000 protein以上を同定するのはかなりのノウハウが必要と感じます。
【要約】この研究では、人間大腸オルガノイドを大腸内に移植(orthotopic transplantation)することで、in vitro培養されたオルガノイドがin vivoに近い形質を獲得するかを、Deep Visual Proteomics(DVP)という空間的・細胞内プロテオーム解析法を用いて調べています。DVP により、組織切片から細胞種を画像で識別し、その後対応するタンパク質を質量分析で定量することが可能です。移植したオルガノイドは、未移植のものと比べて上皮分化マーカー(例:KRT20など)をはじめ、分泌蛋白、代謝関連蛋白、細胞接着/構造蛋白など多くのプロファイルで、正常な大腸上皮や腫瘍組織の in vivo 表現型により近づくことが示されました。また、免疫細胞などの周囲微小環境との相互作用も促進され、in vitroでは見られない特徴が観察されています。結論として、orthotopically transplantedオルガノイドは培養系よりも自然な大腸の生理・構造をモデル化する能力が高く、疾患モデリングや創薬におけるより信頼性の高いプラットフォームとして期待されます。
https://www.cell.com/cell-systems/fulltext/S2405-4712(25)00229-7

コメントを残す